
产品中心
PRODUCT

电 话:0898-08980898
手 机:13877778888
联系人:xxx
E_mail:admin@Your website.com
地 址:广东省清远市
因为一个“很难很重要的问题”我从计算机转向研究蛋白质
2006到2014年期间,我开始使用机器学习去研究这个问题。传统的机器学习方法是直接把蛋白质的氨基酸序列映射到一个三维构型上去,比基于物理或是统计的方法做得好一点点,但也还有很多问题。当时大家认为这个问题没办法做出来◆★★,期间很多人都离开这个领域;另外,由于这个问题很长时间都没有得到什么本质上的改变,所以申请研究经费也非常困难。



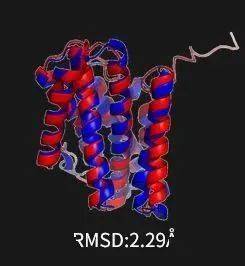
这是我们当时预测的一个有两百多个氨基酸的膜蛋白的结构,误差大概是2■★◆.29个埃◆★★■,已经非常接近用实验技术解出来的结构的分辨率了。
那什么是深度学习◆◆?它其实是模拟大脑神经元的工作方式来进行预测,好处在于不需要告诉计算机怎么一步步去做★◆★★◆,只用给计算机输入和输出。也就是说,我们只要给计算机氨基酸序列,告诉计算机它们对应的一些真实结构或者实验结构,就可以教计算机自主学会预测蛋白质的结构。
我本科和硕士学的是计算机,研究计算机算法与计算机网络,以及分布式系统以及计算机安全■◆★★★,其实跟生物没有什么关系◆★◆◆。
比如说,我们能不能预测蛋白质跟其他分子的相互作用★■★■★★,这跟蛋白质的功能预测★◆◆、跟制药息息相关,因为蛋白质在细胞里面执行功能是通过跟其他分子结合在一起去实现的,所以这是一个非常重要的问题◆★■★◆。另外■★◆◆■,我想这几年大家对抗体已经比较熟悉了。当设计出一个抗体之后★★★,我们可以预测这个抗体跟抗原到底怎么结合■★◆◆,通过这种方式我们可以去估计这个抗体到底有多好◆★★■★■。最后,我们也可以用人工智能去设计自然界不存在的蛋白,这些蛋白可以用来制药,也可以用在工业生产上★★◆■,比如我们可以设计一些新的酶来提高工业催化的效率。


这张幻灯片的左边展示了四个不同蛋白的结构,很漂亮,我们用软件把它画成了卡通形式◆◆◆,简化了结构的复杂度。但在右边,我们把蛋白质复合物的原子都画出来了■★◆★■,就变得非常复杂。我们可以用不同软件、不同表示形式把蛋白质的三维结构给画出来◆■■,去观察蛋白质到底长什么样◆★◆◆■。根据蛋白质的结构,我们可以在原子层面去分析■◆★、去预测这个蛋白质它到底会执行什么样的功能■◆。
大概是读博士一年半的时候■★★,一个偶然的机会我接触到生物学★★◆◆★,接触到蛋白质结构预测问题◆◆★。我当时的想法很单纯,觉得这个问题很有趣,也非常重要,并且非常难,所以就想去研究它。当我们选择研究课题的时候,通常要选择一些比较重要和困难的问题,因为如果这个问题不重要的话,就白花时间去研究了★★■,没有人在乎■◆;如果这个问题不那么难◆◆■,可能几年之内就被别人做出来了,其实也没有多大意义★◆。
演讲嘉宾许锦波:《AI预测蛋白质结构◆◆,但这只是一个开始》 拍摄:Vphoto

原子在细胞里面有相互作用力,最后会形成一个比较稳定的状态去执行某种特殊的功能。所以,虽然我们对蛋白质的分子式已经很了解,但知道这些组成蛋白质的原子最后会形成怎么样的构型仍是个很困难的问题。
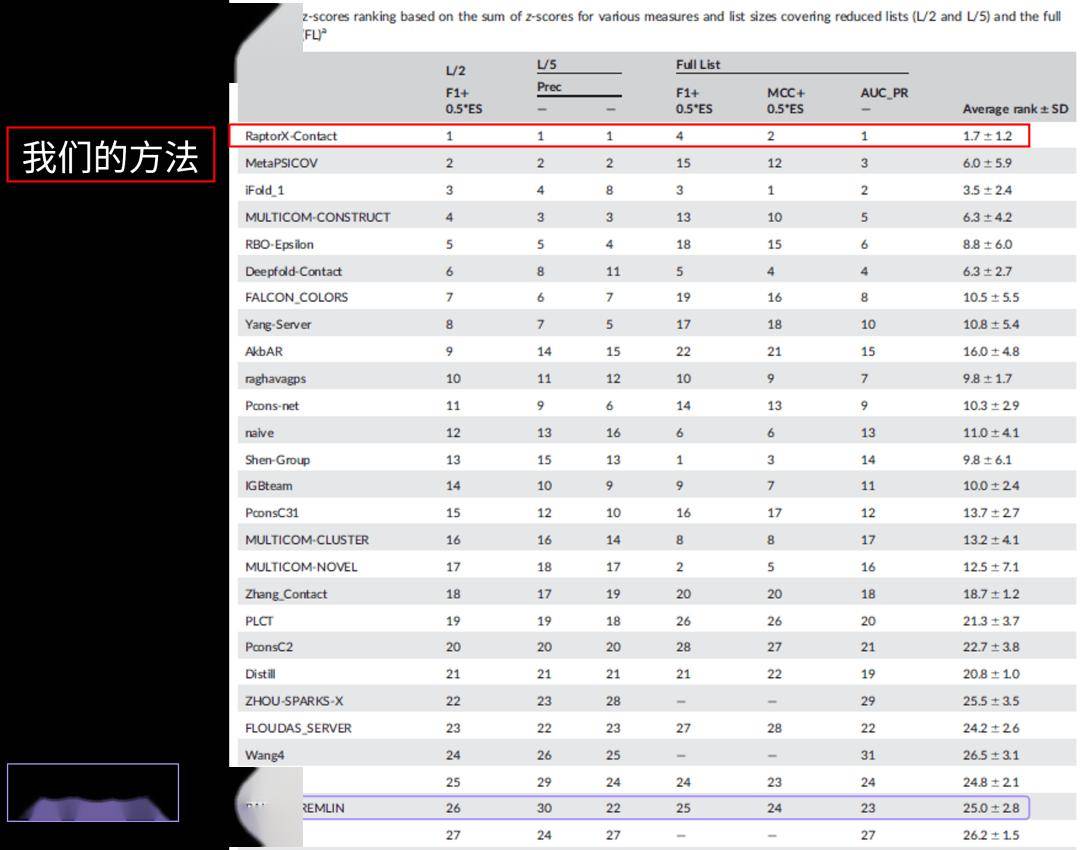
2016年暑假,我们参加了全球蛋白质结构预测比赛(CASP)。虽然这时候我们还没有把方法完全实现好,但在测试中我们的算法已经是排名最好的,远远好于传统的统计方法。

我们当时的方法叫★■★■◆◆“深度卷积残差神经网络”。预测思路是,首先预测蛋白质里面两个氨基酸在空间中是靠得比较近还是离得比较远◆◆◆★,再把它们的三维坐标重构出来。2016年暑假,我们发现这个方法可以大幅度地提高蛋白质三维结构预测精度,在那年秋天写成一篇论文贴到网上去,引起了领域内很多人的关注。
2001到2006年★★◆,我主要使用能量优化的方法去做蛋白质预测。大家普遍认为蛋白质会折叠到最小能量状态■◆★◆,如果我们能把某个蛋白质的能量最优化■■★◆■◆,理论上就可以算出它的结构。这种方法会教计算机一步步怎么去优化能量,从而达到预测蛋白质结构的目的■◆◆。当时虽然取得了一定成果◆◆★★■,但结果还是无法令人满意■■★■◆,预测出来的结构离实验技术测出来的非常远◆★★◆★。于是我意识到能量优化方法是走不通的。
我们知道■◆◆★,细胞里有三种大分子■◆◆:DNA、RNA和蛋白质。DNA和RNA记录了我们的遗传信息,但真正在细胞里面执行功能的是蛋白质。蛋白质怎么去执行功能?它在细胞里面折叠成固定的三维构型,这个三维结构决定了它的功能,这也正是为什么我们想研究蛋白质的三维结构★★◆■◆◆。
2014年◆◆★★◆,我们设计了一种新的深度学习算法,开始使用深度学习去研究蛋白质结构■★。我们先在蛋白质二级结构预测上测试,发现深度学习对这个简单问题有效,就激发了我们去做进一步的研究。
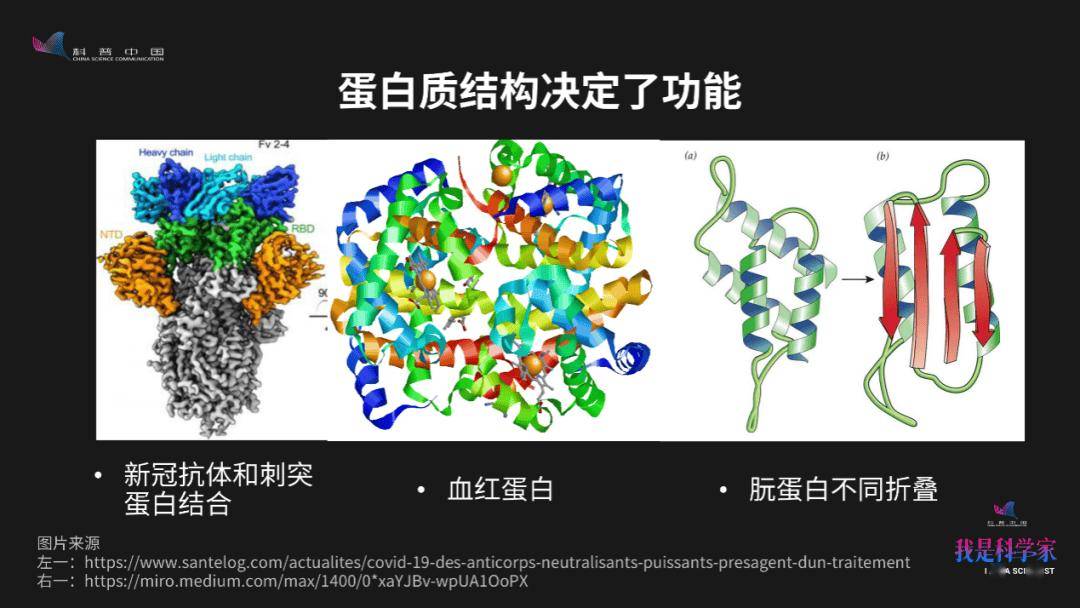
这里展示了蛋白质结构与功能的三个例子。左边这幅图显示了新冠抗体和病毒刺突蛋白结合的样子◆★★★★■,抗体通过和刺突蛋白结合,就可以阻挡新冠病毒进入人体细胞。中间这幅图片是一个血红蛋白,它的主要功能是帮助我们把氧气运输到人体各个地方■★★★◆。怎么运输?你看到中间有些小球,这些小球表示的是铁原子◆◆■■■★。血红蛋白把铁原子包裹起来◆★■★◆,铁原子跟氧气结合,通过这种方式把氧气运输到各个细胞里面去★■★。最右边的蛋白(朊蛋白)跟疯牛病有关系,这里显示了朊蛋白两种不同构型,左边的构型是正常的折叠状态,右边的是不正常的折叠状态。如果朊蛋白的折叠是右边这个形状的话,就会引起疯牛病。
2020年◆★★★■,DeepMind继承了我们的方法■◆,开发了新一代的深度学习方法★◆★。那一年有很多人知道人工智能已经颠覆了蛋白质结构预测,这种颠覆带来的改变非常大,特别是改变了分子生物学家做研究的范式——以前分子生物学家研究一个蛋白质◆◆◆★★,都是基于氨基酸序列去研究蛋白质的功能,但现在我们有了精确的结构预测★◆◆■◆,分子学家可以直接基于预测出的结构去研究蛋白质的功能★◆。
在以前,我们没有特别好的方法去确定这些原子在三维空间中到底会处于什么样的位置★◆◆★。
那时候,科学家已经研究蛋白质结构这个问题大概四十年了★★■■■◆,它真的是个非常难的问题。刚开始研究的时候■■★◆,我也碰到很多困难。比如,刚才也说了◆■★◆,我本身没有学过生物学,所以前半年我读那些专业论文,很多专业名词都不知道是什么意思,也没有人去问,因为我的博士导师当时在加州学术休假★■★■★,基本上两个月才能见到他一次★■;而且那时候也没有现在这么方便的网络视频、网络会议,所以刚开始的时候非常困难◆◆。
于是■■■◆,我就从一个传统的计算机研究者切换到一个计算生物学研究者,想在读博士期间花几年时间去研究这个问题■★■◆■。当时根本没有想过自己以前根本没有学过生物学,心想如果一点结果都做不出来也没关系,到时候大不了去硅谷写代码,当码农。
首先,这些方法需要非常长的时间去测定哪怕一个蛋白质的三维构型◆■★■◆◆。另外■■★★■,它们的费用非常高◆■。更致命的是,并不是所有蛋白质的三维构型都可以用这些实验技术给测出来。所以,我们需要另外想出方法去获得定蛋白质的三维构型◆■,这也是为什么我们想做蛋白质结构预测——我们想通过计算技术去把蛋白质的三维结构给算出来★■★★★,而不是用实验仪器测出来■◆★。
2012年★■,深度学习开始在图像识别领域展示出威力◆◆,就有人很自然地想到,我们能不能用深度学习去做蛋白质结构预测■★★。然而当时他们得到的结果跟传统的机器学习方法没有任何区别■★★,也就是说在这个领域,最初得到的是个否定的结果。
所以,2019年和2020年,我又进一步去发展了我们这个方法,实现了不使用共进化信息去做蛋白质结构预测,预测结果跟实验测出来的结构非常吻合。

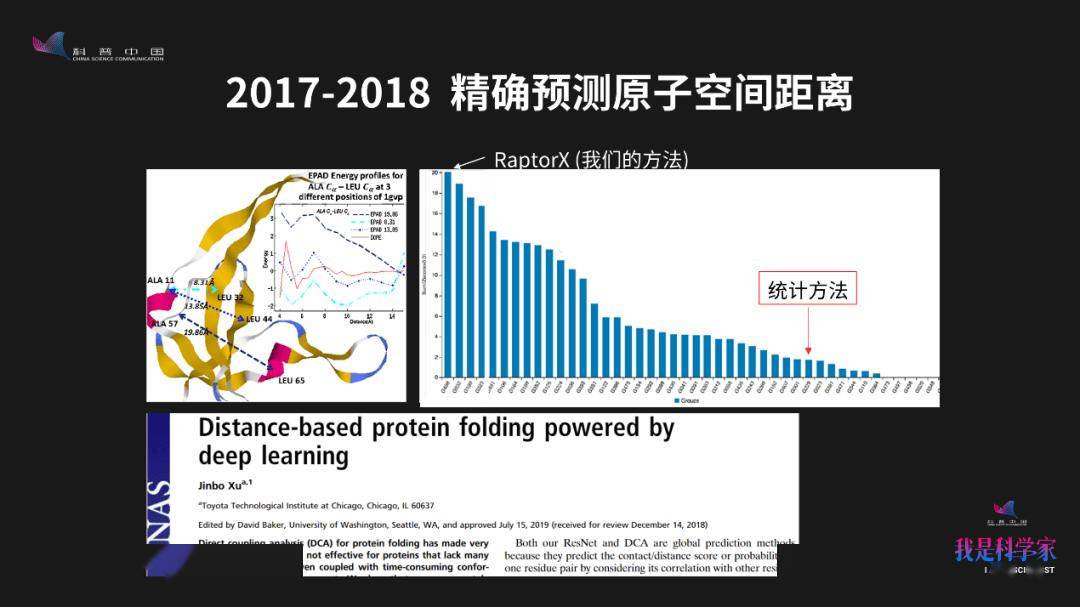
这篇论文在2017年1月份正式发表,在2018年上半年拿到了国际计算生物学的旗舰期刊PLoS Computational Biology的创新突破奖。
我们2016年和2018年的算法都需要利用蛋白质的共进化信息■★★。什么叫共进化信息?假如两个氨基酸在空间中靠得比较近,那么在进化过程中,它们就可能会同时进化■◆◆,这种现象就叫共进化★■■◆★★。但现实中,有些情况下蛋白质是没有共进化信息的,一种是人工设计的蛋白★■◆★■◆,它不是自然界存在的,也就没有共进化信息■★■■■◆;另外一种情况就是蛋白质复合物,如果我们想预测两个蛋白质在空间中怎么结合★★◆◆,很多情况下是得不到两个蛋白质之间的共进化信息。所以我们需要能够在不使用共进化信息的情况下去预测蛋白质结构。
自从人工智能在预测蛋白质获得突破之后,国内也有很多组去研究这个问题,但很多是在重复实现已有的人工智能算法——当然这些工作需要我们去做,但这并不是最好的途径,因为这个领域内还有非常多的问题没有解决★◆◆■★。
2015年和2016年,我们开发了一种更好的深度学习算法,它可以直接用来预测蛋白质的三维结构★■★。
2016年前,当我们谈论蛋白质结构预测◆★★◆■,通常意味着需要非常多的计算资源◆◆。那时候的科学家们通常用超级计算机去做蛋白质结构预测◆★■★■◆,只有少数几个研究组能够真正做到◆★◆■。即使这样,对于很小的蛋白质,预测成功率也非常低。

的其中一半★★■◆■,授予了戴密斯·哈萨比斯(Demis Hassabis)与约翰·朱默帕(John M◆■. Jumper),以表彰他们对

作为计算生物学家,我主要工作是用计算技术去研究生物学问题◆■,所以计算机是我最常用的工具◆◆■。
在2020年★■,人工智能预测蛋白质结构入选了《科学》杂志评出来的“十大科学突破”;到2021年又被《科学》杂志评为“十大科学突破之首■◆★◆”;在今年被《麻省理工科技评论》评为“十大突破性技术★◆”。
北京大学BIOPIC访问教授、北京分子之心科技有限公司首席科学家许锦波,曾在“科普中国-我是科学家”的演讲现场带来演讲《AI预测蛋白质结构,但这只是一个开始》。以下为演讲实录◆◆★★。
怎样才能得到蛋白质结构?在过去的几十年中,科学家们开发了不同的实验技术去观察和测定蛋白质在细胞里的三维构型■◆,其中最精确的一种叫做晶体衍射技术■■◆◆。后续科学家们也开发了另外两种技术,一种是核磁共振,另一种是现在非常流行的冷冻电镜技术■◆■■◆。这三种技术都可以以不同的方式去观察和测定蛋白质的三维构型★■★◆,但也都存在问题。
我本人从2001年开始研究这个问题,我的研究历程大概可以分为三个阶段——
2017年和2018年,我们进一步推广了这个算法◆■★■,把之前预测“氨基酸靠得比较近还是离得比较远”推广到预测两个原子在空间中的距离★◆■★★。比如说,我们可以预测蛋白质里面两个原子在空间中的距离是5埃★◆◆,还是6埃, 7埃。根据预测出的距离★■◆,我们可以把三维坐标重构出来■★。后来我们把这个想法写成一篇论文,发表在2019年《美国国家科学院院刊》上面。
计算机预测蛋白质结构这个问题本身的定义很简单:假设我们有了一个蛋白质的氨基酸序列■◆◆◆■,能不能把它输入到计算机里面去■■★,让计算机输出每个原子的三维坐标■★★?然而,实际操作中是非常困难的◆◆■◆,到目前为止已经研究了将近六十年了。
蛋白质由很多氨基酸通过化学键串在一起。这里我用一些小球去表示这些氨基酸,每种颜色的小球表示一种氨基酸。从数学角度◆◆◆◆,你也可以用一个字符串去表示一个蛋白质的氨基酸序列。每个氨基酸都是由几十个原子形成的,在这页幻灯片的下半部分★■■◆◆◆,我画了一些原子结构■■◆■★★,大家可以对蛋白质的分子式有一个直观的认识。现在我们只要知道氨基酸序列,就能知道蛋白质的分子式。
演讲嘉宾许锦波:《AI预测蛋白质结构,但这只是一个开始》 拍摄:Vphoto
Copyright © 2012-2018 贝斯特游戏官方网站,贝斯特全球最奢游戏官网,贝斯特全球最奢游戏60年网站 版权所有
电 话:0898-08980898 手 机:13877778888 传 真:0000-0000-00 E-mail:admin@Your website.com
地 址:广东省清远市

扫码关注我们